
Summary
El haplogrupo N o macrohaplogrupo N* es un haplogrupo mitocondrial humano cuyos descendientes están esparcidos por todos los continentes. Cuenta entre sus haplogrupos derivados a A, S, I, W, X, Y, R* y varios subgrupos de N.
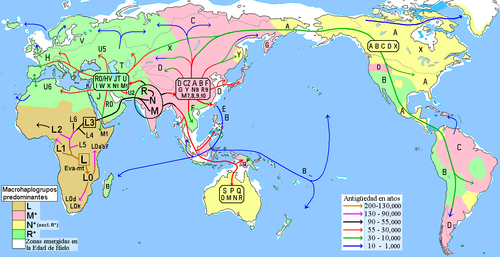
N se habría originado en algún punto de la ruta migratoria costera del Sur de Asia hace unos 65.000 años y está definido por los marcadores genéticos 8701, 9540, 10398, 10873 y 15301.
Origen y dispersión
editarComparte con el haplogrupo M similitudes o paralelismo en su origen y en el proceso migratorio. M y N son clados próximos derivados del haplogrupo L3, por lo que están estrechamente relacionados con la expansión de la humanidad fuera de África. Al igual que M tiene una antigüedad aproximada de 60.000 a 65.000 años[1] y un origen probable en Asia Meridional, dada la importancia de esta región en el proceso colonizador temprano fuera de África.
Subgrupos de N como N1 y otros son encontrados en el Cercano Oriente, ya sea por temprana divergencia en la ruta de África o por subsecuentes migraciones de regreso hacia Eurasia Occidental.[2] En la medida de sus frecuencias, N es considerado un haplogrupo euroasiático-occidental con su centro más importante de expansión en el Cercano Oriente,[3] sin embargo se extiende también desde sus orígenes hacia el Sudeste de Asia, Asia Oriental y las regiones más remotas como Australia, Siberia y finalmente América.
Grupos derivados y su distribución
editarN* se encuentra presente en todos los continentes. En Asia está la mayor diversificación. A través del macrohaplogrupo R* alcanza en Europa las más altas frecuencias y gran difusión en los demás continentes. Haplogrupos derivados importantes son A y X en América, S en Australia, A e Y en Siberia y en menor proporción I, W y X en Europa.
El haplogrupo N y sus descendientes se relacionan de la siguiente manera:[4]
- Haplogrupo N (8701, 9540, 10398, 10873, 15301)
- N1'5 (1719)
- N5: (5063) Encontrado en la India.[5]
- N1 (10238, 12501): Bien extendido en Eurasia Occidental.
- N1b: Encontrado en judíos askenazí y Medio Oriente, 3% en el Levante, Arabia y Egipto.[6] También en Irán, Pakistán (en Makrán 9.5%),[7] el Cáucaso y poco en Europa Occidental.
- N1a'c'd'e'I
- N1c: Encontrado en Polonia[8] y Medio Oriente.
- N1a'd'e'I
- N1d: En India[5] y Pakistán.
- N1a'e'I
- N1a: Bajas frecuencias en la península arábiga, Kenia, Etiopía y Egipto.[9] Igualmente raro en la región del Volga y Sur de los Urales en Asia Central.[10] Sin embargo, antiguas poblaciones de Europa de hace 2.500-10.000 años[11] y de Asia Central relacionadas con los escitas, tuvieron frecuencias de N1a mucho más importantes.
- N1e'I
- N2 (189, 709, 5046, 11674, 12414): Propio de Eurasia Occidental.
- N2a: Poco en Europa Occidental (0.25%).[12]
- W: Muy extendido pero en bajas frecuencias en Europa, Medio Oriente y Asia Meridional.
- N8: Encontrado en Guangxi (China).[13]
- N9 (5417)
- N9a: Común en China (en Qinghai 8%),[14] también en Indochina, en sondaneses y en Asia Central (en Uzbekistán 7%).[7]
- N9b: En el Japón.
- Y: Típico de Siberia Oriental, en especial en los nivjis.[15] También presente en Asia Oriental y Filipinas.
- N10: Disperso en China y el Sureste asiático.[13]
- N11
- N11a: En China.
- N11b: En Filipinas.[16]
- N13: En Australia.[17][1]
- N14: En Australia.[18]
- N21: En malayos de Malasia e Indonesia, especialmente en los aborígenes semelai (31%).[19]
- N22: En Sumba (Indonesia) con 6%,[20] en la península Malaya y en Mindanao (Filipinas).[21]
- A: Típico entre los amerindios, predomina especialmente en América del Norte. Se encuentra también en Asia Oriental, Central y en pueblos de Siberia como los chukchis.[15]
- O o N12 (6755, 9140, 16213): Encontrado en la región central de Australia[17] y en la isla Flores (Indonesia).[22]
- S: Difundido ampliamente entre los aborígenes australianos.[23]
- X: Muy disperso pero en bajas frecuencias en todo Eurasia Occidental, con la mayor incidencia tanto en X1 como en X2 en drusos de Israel (27%).[24]
- X1: En África del Norte y Cercano Oriente.
- X2: Muy extendido en Europa, Norte de África, Medio Oriente, Cáucaso, Asia Central, raro en Siberia y llega hasta América, en especial en los nativos algonquinos del Canadá.[25]
- Haplogrupo R*: Macrohaplogrupo de gran diversificación, extensión y predominio en Eurasia Occidental.
- N1'5 (1719)
Véase también
editar| Eva mitocondrial (L) | |||||||||||||||||||||||||||||||||||||||
| L0 | L1–6 | ||||||||||||||||||||||||||||||||||||||
| L1 | L2 | L3 | L4 | L5 | L6 | ||||||||||||||||||||||||||||||||||
| M | N | ||||||||||||||||||||||||||||||||||||||
| CZ | D | E | G | Q | A | I | O | R | S | W | X | Y | |||||||||||||||||||||||||||
| C | Z | B | F | R0 | R2'JT | P | U | ||||||||||||||||||||||||||||||||
| HV | JT | K | |||||||||||||||||||||||||||||||||||||
| H | V | J | T | ||||||||||||||||||||||||||||||||||||
Enlaces externos
editar- Árbol filogenético de N de van Oven M & Kayser M. 2009
- The Haplogroup N mtDNA Study de Family Tree DNA.
- Phylogeny of Macrohaplogroup N, de American Society of Human Genetics
Referencias
editar- ↑ Vincent Macaulay et al. 2005, Single, Rapid Coastal Settlement of Asia Revealed by Analysis of Complete Mitochondrial Genomes, Science 1034-1036 DOI: 10.1126/science.1109792
- ↑ Family Tree DNA The Haplogroup N mtDNA Study
- ↑ Spencer Wells 2006, Deep Ancestry: Inside the Genographic Project Archivado el 24 de junio de 2009 en Wayback Machine., National Geographic.
- ↑ van Oven M, Kayser M. 2009. Updated comprehensive phylogenetic tree of global human mitochondrial DNA variation. Hum Mutat 30(2):E386-E394. http://www.phylotree.org, mtDNA tree Build 2 (14 Oct 2008)
- ↑ a b Malliya gounder Palanichamy et al. 2004, Phylogeny of Mitochondrial DNA Macrohaplogroup N in India, Based on Complete Sequencing: Implications for the Peopling of South Asia, Am J Hum Genet. 2004 December; 75(6): 966–978.
- ↑ Doron M. Behar et al. 2006, The Matrilineal Ancestry of Ashkenazi Jewry: Portrait of a Recent Founder Event Archivado el 2 de diciembre de 2007 en Wayback Machine., Am. J. Hum. Genet. 2006;78:000–000.
- ↑ a b Lluís Quintana-Murci et al. 2004, Where West Meets East: The Complex mtDNA Landscape of the Southwest and Central Asian Corridor. Archivado el 23 de noviembre de 2009 en Wayback Machine. Am. J. Hum. Genet. 74:000–000, 2004
- ↑ B. A. Malyarchuk et al.2002, Mitochondrial DNA variability in Poles and Russians Archivado el 17 de septiembre de 2012 en Wayback Machine., Ann. Hum. Genet. (2002), 66, 261-283, University College London.
- ↑ Abu-Amero et al. 2008 February. "Mitochondrial DNA structure in the Arabian Peninsula", BMC Evolutionary Biology. 8(45): 52.
- ↑ a b Miroslava Derenko 2007 Phylogeographic Analysis of Mitochondrial DNA in Northern Asian Populations The American Journal of Human Genetics 81, 5, Nov 07, 1025-1041
- ↑ Ellen Levy-Coffman 2006 We Are Not Our Ancestors: Evidence for Discontinuity between Prehistoric and Modern Europeans Archivado el 8 de abril de 2009 en Wayback Machine. Journal of Genetic Genealogy Dic 08
- ↑ Turchi, Chiara et al.2008, Italian mitochondrial DNA database: results of a collaborative exercise and proficiency testing (enlace roto disponible en Internet Archive; véase el historial, la primera versión y la última)., International Journal of Legal Medicine, 122, Nº3, May 08, 199-204(6)
- ↑ a b Kong, Qing-Peng et al 2011, Large-Scale mtDNA Screening Reveals a Surprising Matrilineal Complexity in East Asia and Its Implications to the Peopling of the Region.
- ↑ Yong-Gang Yao 2001 Phylogeographic Differentiation of Mitochondrial DNA in Han Chinese Am J Hum Genet. 2002 March; 70(3): 635–651
- ↑ a b Yelena B. Starikovskaya 1998, mtDNA Diversity in Chukchi and Siberian Eskimos: Implications for the Genetic History of Ancient Beringia and the Peopling of the New World The American Journal of Human Genetics 63, 5, Nov 98, 1473-1491
- ↑ Gunnarsdóttir, Ellen et al 2010, High-throughput sequencing of complete human mtDNA genomes from the Philippines
- ↑ a b Georgi Hudjashov et al. 2007, Revealing the prehistoric settlement of Australia by Y chromosome and mtDNA analysis, PNAS May 22, 2007 104 21 8726-8730.
- ↑ Melanie J. Pierson et al. 2006, Deciphering Past Human Population Movements in Oceania: Provably Optimal Trees of 127 mtDNA Genomes Archivado el 23 de noviembre de 2009 en Wayback Machine., MBE Advance Access published July 19, 2006.
- ↑ Catherine Hill et al. 2006, Phylogeography and Ethnogenesis of Aboriginal Southeast Asians, Mol. Biol. Evol. 23(12):2480–2491. 2006 doi:10.1093/molbev/msl124.
- ↑ C. Hill et al. 2007, A Mitochondrial Stratigraphy for Island Southeast Asia. Am J Hum Genet. 2007 January; 80(1): 29–43.
- ↑ Tabbada, Kristina. et al. 2009, Philippine mitochondrial DNA diversity: a populated viaduct between Taiwan and Indonesia? Archivado el 23 de noviembre de 2009 en Wayback Machine. Molecular Biology and Evolution, doi:10.1093/molbev/msp215
- ↑ Stefano Mona et al. 2009, Genetic admixture history of eastern Indonesia as revealed by Y-chromosome and mitochondrial DNA analysis (enlace roto disponible en Internet Archive; véase el historial, la primera versión y la última)., Molecular Biology and Evolution, doi:10.1093/molbev/msp097.
- ↑ van Holst Pellekaan SM 2006, Mitochondrial genomics identifies major haplogroups in Aboriginal Australians. Am J Phys Anthropol. Oct 06;131(2):282-94
- ↑ Reidla, Maere et al. 2003, Origin and Diffusion of mtDNA Haplogroup X.
- ↑ Dolan DNA Learning Center - Native American haplogroups: European lineage, Douglas Wallace


